2018-10-01
上一期为大家介绍了过去一年里CRISPR技术在动物造模及单碱基技术方面取得的重大突破。本期继续为大家从功能基因组筛选、细胞谱系示踪及疾病诊断方面谈谈CRISPR-Cas系统的技术运用。
一、大规模基因功能的筛选
尽管测序和基因组编辑技术取得了重大进展,但是解析复杂的基因型-表型关系仍然是数量遗传学的一个主要障碍。作为强大的基因编辑工具,CRISPR-Cas9系统能够在蛋白编码基因的外显子区域产生移码突变而彻底破坏蛋白表达及功能,这一特性被广泛应用于基因的大规模功能性筛选研究,这也将大大加速评估基因变异的功能影响。
1. 利用CRISPRa构建无偏向性的耐药相关LncRNA筛选平台
2018年4月Cell期刊上刊登一项研究开发了一种开创性的方法来鉴定和确定在急性髓细胞白血病(AML)中lncRNA在化疗药物产生耐药性中所起的功能作用。这一技术通过结合公开可获得的药理学数据库的信息与CRISPR技术,筛选影响治疗反应的编码基因和非编码基因。作为一种全基因组筛查平台,既不偏向于编码基因,也不偏向于非编码基因,能够筛选到新的治疗靶标[1]。

2. 利用一一配对的靶点-模板策略实现全基因组高效精准突变
理解DNA突变体的功能效应对于基础生物学、进化生物学和医学遗传学的研究至关重要;尽管CRISPR-Cas9技术可以实现体内数以千计细胞发生多基因诱导突变的可能,然而高通量测序这些突变体的特异性仍然是技术的瓶颈。2018年4月Nat Genet期刊刊登了一篇研究,通过改造CRISPR-Cas9系统,解决了目前高通量测序数以万计基因组编辑结果的这一壁垒,开发了一种基于CRISPR的全基因组高效精确突变工程的方法。如图所示,通过构建10000对编码gRNA靶向序列及其相应的顺式修复模板的质粒文库来实现靶点和模板的一一配对,将文库递送到酵母细胞中监测基因突变的影响,突变效率高达95 %。这一策略可以高效精确地追踪大量基因突变对细胞功能的影响,为研究基因的功能提供了强有力的分析工具[2]。
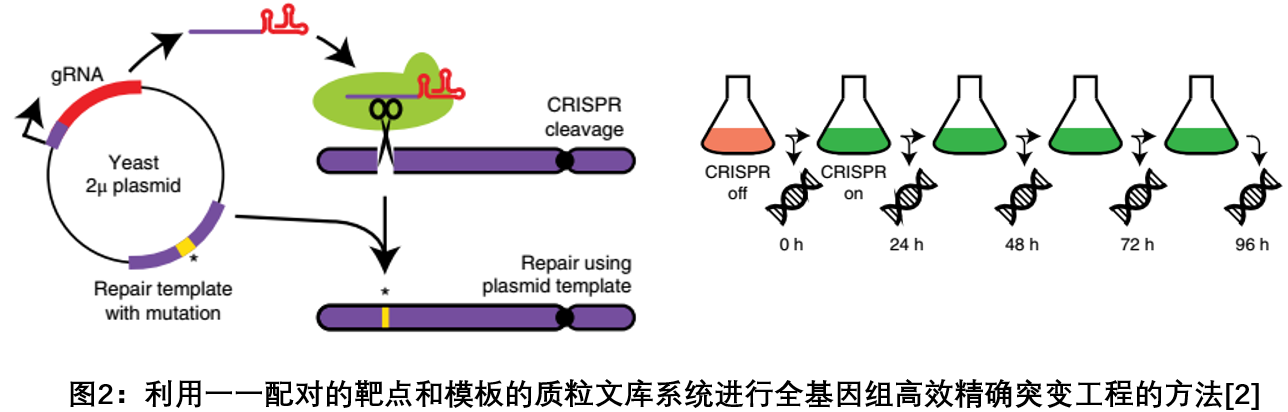
3. Guide+donor:利用CRISPR-Cas9在酵母中高通量构建和功能分析DNA序列变异文库
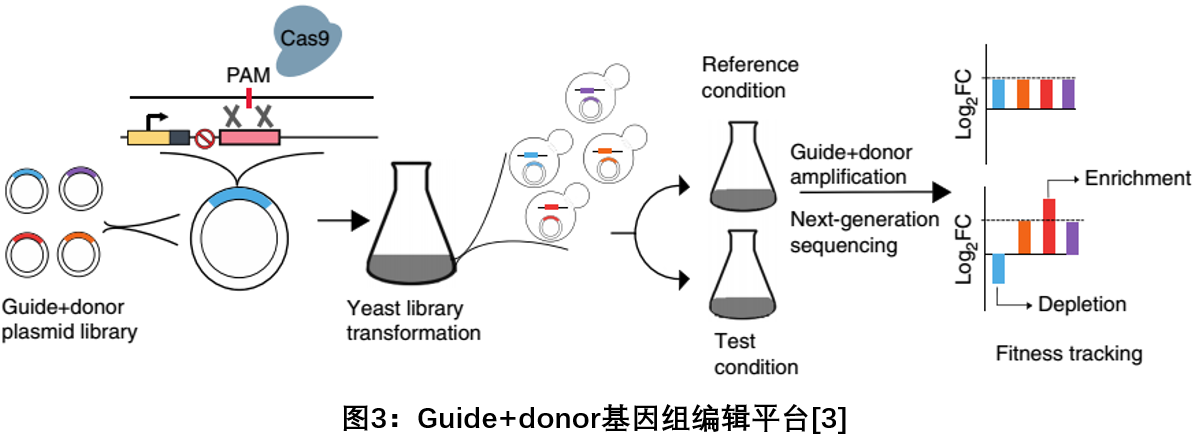
2018年5月,哈佛基因编辑大牛Church研究组在Nature Biotechnology发文,介绍了一种基于Cas9的方法,在酵母中高效(80-100%)产生特定遗传变异(缺失、替换和插入)的突变体文库,以及整体跟踪其适应性的方法。使用guide+donor方法精确地剔除了酵母基因组中的315个基因,并评估和鉴定了对细胞适应性存活中起重要作用的基因。新的方法不仅能以高通量的方式在酵母菌中精确地进行功能基因组研究,还能深入挖掘低频基因突变及非编码序列的基因功能,为基因功能分析打开了新的大门[3]。
4. CHAnGE:一种基于CRISPR-Cas9和同源定向修复相关的大规模基因组筛选方法
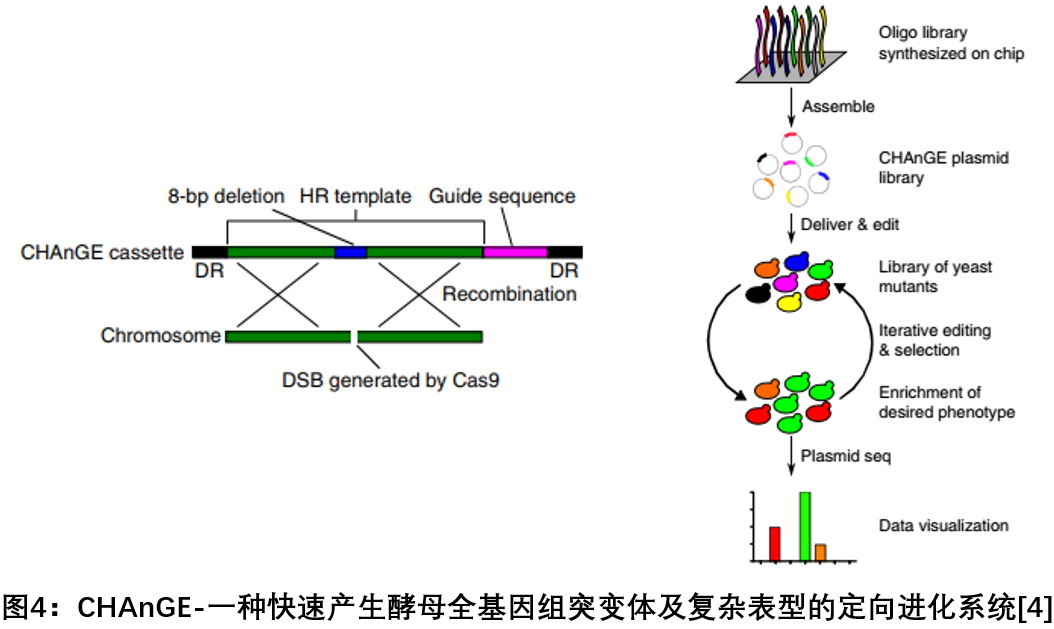
2018年7月,美国伊利诺伊大学赵惠民研究组在Nature Biotechnology发文,开发了一种基于CRISPR - Cas9和同源定向修复相关的大规模基因组筛选方法CHAnGE,可以在全基因组范围快速产生成千上万种精确的酵母单核苷酸突变体,追踪基因突变对细胞的影响,突变效率超过98 %。该研究通过创建全基因组基因突变集合验证了该方法对单核苷酸分辨率基因组编辑的可行性,解决了之前的方法编辑效率局限性的问题。该方法能够对酿酒酵母进行全基因组规模的快速工程化,并进行精确和可追踪的基因突变[4]。
5. MAGESTIC:一种在酵母中可追踪基因组条形码的多重精确基因组编辑技术
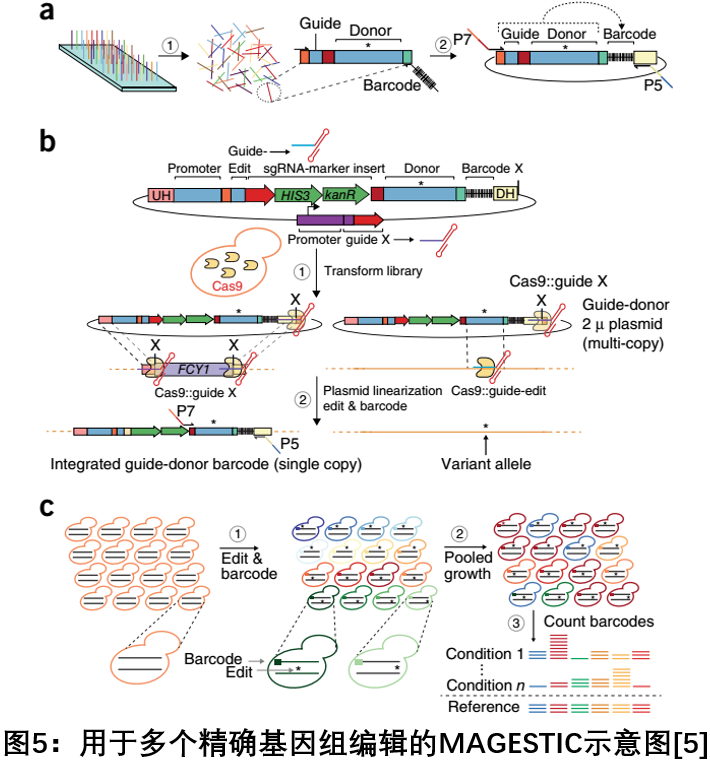
2018年7月,美国斯坦福大学的 Steinmetz LM课题组在Nature Biotechnology发文,开发了一种基于Cas9技术利用短的、可追踪的整合性细胞条形码(MAGESTIC)在酿酒酵母中进行多重精确基因组编辑的方法。MAGESTIC使用合成的阵列gRNA-donor的寡核苷酸的质粒系统进行高通量编辑,具有基因组条形码整合的功能,可以防止质粒条形码的丢失,维持稳定的表型。利用LexA - FKH1P融合系统将供体DNA主动招募到DNA断裂部位的方法,可以提高同源重组的效率。并且相比donor融合到Cas9的方式具有更大的优势,一方面可以招募更多拷贝的donor,同时还不依赖于Cas9与DSBs的持续关联。这一技术克服了目前可行方法的几个限制,即质粒条形码的不稳定性,以及在表型分析期间无法区分靶向RNA和供体模板中的Oligo合成和PCR或测序引起的错误。MAGESTIC技术将广泛用于揭示酵母表型的遗传基础[5]。
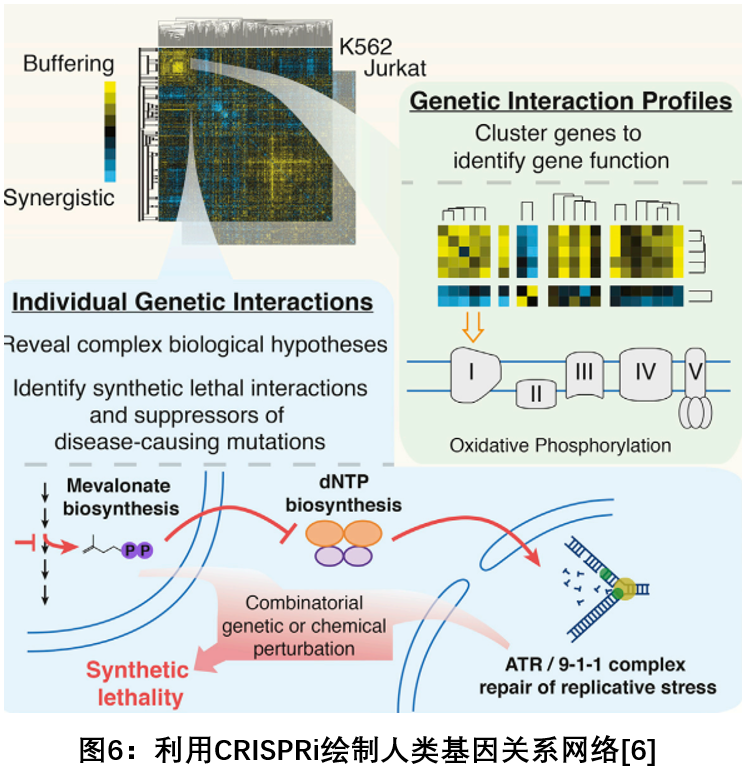
6. 基于CRISPRi的高通量技术快速绘制人类基因的功能图谱
2018年7月,Cell刊登了美国加州大学旧金山分校的研究小组的研究成果,开发了一种基于CRISPR的高通量技术快速地绘制人细胞中将近500个基因的功能图谱,其中的许多基因之前从未被详细地研究过。人类目前研究过的基因还不到10%,剩余的基因功能我们还一无所知。传统测试基因功能所需的方法既昂贵又耗时。开发一种快速而又全面地确定这些未经研究的基因功能将有助于科学家对生物学的更广泛理解。该研究使用了一种被称作基因相互作用图谱(genetic interaction mapping)的技术,用于建立对人体中基因功能的全面解析。利用一种被称作CRISPR抑制(CRISPRi)的工具,能够在不编辑DNA本身的情况下降低基因活性,系统性地关闭单个细胞中的成对基因并测量细胞如何作出反应,阐释这两个基因之间的关系,能够快速地归类未知功能的基因。利用这一系统,研究人员鉴定出了参与细胞能量产生的新基因,并解释了为何一些胆固醇药物可用于治疗骨质疏松症而相关药物没有这种效果的长期谜团。该研究最终的目的将计划绘制整个人类基因组中的全基因功能框架[6]。
7. EvolvR:一种结合CRISPR和DNA聚合酶来促进细胞内特定基因进化的平台
2018年8月,加州大学伯克利分校创新基因组学研究所在Nature上发文,提出了一种可以利用自然进化力量的变革性新方法---一个促进细胞内特定基因进化的平台。他们将这个新系统命名为“EvolvR”,这一系统是将Cas9同一种DNA聚合酶融合,利用一种nicking-Cas9只切割两条DNA链中的一条,形成的特殊切口会通过聚合酶补齐。DNA聚合酶补齐的过程会产生错误,导致出现各种不同类型的突变。因此聚合酶这种错误能带来更多的基因多样性,利用EvolvR人为地制造随机突变,创造数百万种不同的序列组合,从中发现许多人为需要的突变物种。这一系统甚至可以实现在一次实验中得到数千种不同的基因多样化,创造出全新的功能,而不仅仅是打开和关闭基因[7]。

8. Pro – Codes:利用蛋白质条形码技术高分辨率分析数百种基因功能的系统
2018年10月,上来自西奈山医院的研究人员在Cell期刊发文,开发了一种在蛋白质水平上的条形码系统( Pro - Codes),使用线性表位的组合来创建更高倍数的蛋白质条形码,可以同时分析数百种基因功能,分辨率可达单细胞水平。利用合成蛋白“抗原决定簇(epitopes)”标记和追踪不同的CRISPRs,Pro-codes能同时兼容数百个CRISPRs以敲除大量基因,并通过质谱流式细胞技术进行分析。目前利用DNA作为条形码的基因筛选技术仅能实现有限的表型和粗略的细胞分辨率。新型Pro-Codes技术能够以单细胞分辨率同时对100个基因进行高维蛋白质水平的表型分析,可以更全面地描述单个基因的生物学效应[8]。

9. 通过CRISPR-Cas9靶向剪切位点来全基因组筛选功能性LncRNA新策略
2018年11月,为弥补之前建立的基因组大片段删除、CRISPRi和CRISPRa等方法进行lncRNA的高通量功能性筛选在效率、质量(假阳性、假阴性)上的不足,北大魏文胜课题组在Nature Biotechnology杂志发文提出了一种新的筛选策略,通过构建特异性靶向基因的剪接位点的新型CRISPR文库,以高通量的方式产生基因的外显子缺失或者内含子滞留。运用这一策略,实现了全基因组水平上对lncRNA功能的高效筛选,筛选并发现了230个lncRNA与细胞存活或增殖相关。并通过进一步分析表明,lncRNA功能在不同细胞种类中具有显著异质性。这一新型高通量技术平台的建立,首次真正实现了从全基因组水平对长链非编码RNA进行基于完全敲除的高通量筛选,该方法学将为系统发现和解析lncRNA功能提供有效工具[9]。
10. SLICE:一种利用Cas9蛋白电穿孔进行单向导RNA慢病毒感染的系统
2018年11月,来自美国加州大学旧金山分校的研究人员在Cell期刊发文,提出一种基于CRISPR的筛选工具-SLICE,一种利用Cas9蛋白电穿孔进行单向导RNA慢病毒感染的系统。基于sgRNA慢病毒文库的传统CRISPR筛选技术因慢病毒载体编码Cas9蛋白时间太长对于体外培养时间有限的原代T细胞来说受到限制,而SLICE系统将通过将sgRNA慢病毒文库与Cas9蛋白电穿孔相结合的方式,能够快速导入患者的原代T细胞中并评估各个基因的功能。这种新方法能够为免疫细胞抗癌的改造以及其他疾病的指导提供强有力的依据[10]。

二、DNA标记与细胞谱系示踪
发育生物学的重点包括构成器官或生物体的细胞类型的多样性以及这些细胞的发育谱系历史。这些方面通常作为一个方向被单独研究,但最近的四篇新论文报道了一种将单细胞RNA测序(scRNA-seq)技术与基于CRISPR的谱系示踪技术相结合来同时解剖转录组细胞表型和谱系历史的方法。近些年来CRISPR-Cas9基因编辑系统已被证明可用于细胞谱系示踪[11]。在经典的实验方案中,条形码阵列被设计到生物体的基因组中;在胚胎发育的早期阶段,胚胎共注射Cas9和导向RNA(gRNA)靶向切割条形码阵列。在CRISPR编辑期间,条形码在不同细胞中以可变的方式被累积切割和修复。随后在特定的发育时间点对条形码序列进行分析,就可以从已有的Cas9诱导的突变类型中推断出细胞之间的谱系关系。
1. 三项标志性研究通过利用scRNA-seq提供的细胞类型信息来研究斑马鱼的发育过程,展示CRISPR系统在细胞谱系示踪中的可行性
l Alemany等人开发了一种被称为ScarTrace的方法,这一方法是在Cas9 蛋白或RNA同靶向GFP的gRNA一起注射胚胎后,靶向八个串联拷贝的GFP基因的转基因基因组簇。对分化后的组织进行scRNA-seq分析,该团队使用一种分选和自动化辅助的转录组测序(SORT-seq)方案。尽管Cas9编辑的GFP序列('伤疤')通常在细胞中表达,因此可以从转录组中读出,但SORT-seq结合了巢式PCR步骤,从单个细胞中的剩余基因组DNA中读出瘢痕序列,从而最大程度地减少缺乏GFP表达的细胞的谱系信息的丢失。研究人员分析了各种组织的克隆历史 - 包括造血系统,成体大脑和眼睛以及再生鳍 - 以提供各种发育信息,例如作为特定分化细胞亚群前体的祖细胞的数量和类型[12]。
l Spanjaard等人则报告了一种概念上同前者类似的谱系追踪方法,通过核酸酶激活来编辑普遍存在的序列(LINNAEUS),让人们能够确定细胞类型和细胞谱系。该方法涉及胚胎注射Cas9蛋白和靶向16-32个遍布基因组的红色荧光蛋白(RFP)基因的整合位点的gRNA。使用基因组分散的靶序列的基本原理是避免附近的阵列拷贝之间的删除,这可能会删除它们之间的含大量信息的拷贝。LINNAEUS适用于斑马鱼幼体,以及成体心脏,肝脏,胰腺和大脑。该团队使用基于液滴的微流体进行scRNA-seq,突变的RFP序列从转录组中读出,并被分成一个用于转录组范围分析的文库和一个靶向分析RFP序列的文库。除了实验过程,Spanjaard等人突出了生物信息学的挑战和对定制分析方法的需求,因为物种进化研究得出的标准系统发育算法对细胞谱系重建来说并不是最理想的,主要是由于需要大量(数万)的单细胞比较和谱系条码不能完整的检测出来[13]。
l 第三项研究中,Raj等人着重探索如何克服胚胎注射Cas9仅在发育的最初几个小时内有效的限制。因此,除了胚胎注射Cas9蛋白和4个靶向RFP条形码的1-4位点的gRNA之外,斑马鱼还同时转基因编码Cas9和另外5个靶向RFP条形码5-9位点的gRNA,这些gRNA均可通过热诱导在特定的时间表达来监测后期发育的阶段。这一方法被称为scGESTALT(一种基于之前报道的GESTALT示踪方法的单细胞衍生技术),scRNA-seq同Spanjaard等人类似,使用基于液滴的微流体技术和转录组的靶向富集来测序条形码。 Raj等人的分析主要集中在大脑上,确定了神经细胞类型之间的许多谱系关系以及发育转变期间可能的基因表达关系[14]。这些强大的细胞示踪技术可用于研究各种正常和病理的发育现象,还可用于其他模式生物。
2. “创始小鼠(founder mouse)”为CRISPR系统在哺乳动物细胞中进行细胞谱系示踪创造可能
上述研究在细胞和低等脊椎动物中证实了CRISPR-Cas9基因编辑系统在细胞谱系示踪中应用的可行性。然而,目前还没有在老鼠身上进行验证,老鼠是一种在许多方面(如发育方面)与人类健康更相关的模式生物。小鼠发育过程较长,以及在其整个发育过程中分化成众多谱系,因而需要持续生成高度多样化的条形码,最大限度地减少不必要的重写事件,以最大限度地提高成功记录重要事件的几率。
在一项新的研究中,来自哈佛大学George Church课题组通过构建了一种含有多个独立的条形码位点分散阵列(hgRNAs)的“创始小鼠”,将创始小鼠与表达Cas9蛋白的小鼠杂交,子代小鼠因为hgRNA会在整个妊娠期随机累积突变,在每个谱系中产生独特的突变,并且不会删除早期的突变,使得相关的细胞具有更加相似的突变谱或条形码。在发育条形码小鼠中,广泛表征每种hgRNA的活性谱和突变等位基因,并在发育早期对谱系树进行自下而上的后期重建,从其根部的第一个分支开始,并追踪细胞的原始谱系。这一研究为哺乳动物模型中的体内条形码和谱系追踪提供了一个可行的通用平台系统[15]。
三、基于CRISPR-Cas系统的疾病诊断技术
快速分子诊断技术在许多领域非常有用,包括公共卫生、环境检测和刑事侦查等。基于CRISPR开发的体外诊断技术入选2018年世界十大科技进展。CRISPR-Cas系统现已被证明可特异性地识别并切割DNA和RNA,这暗示了该系统作为通用核酸识别工具的潜在用途。由于靶序列和CRISPR-Cas系统之间识别的特异性,CRISPR-Cas系统已被用于检测点突变或单核苷酸变体。这项技术能够通过分析病原体的DNA或RNA来检测感染过程中的病原体。下面列举了今年以来CRISPR-Cas系统在分子诊断方面取得的重大突破(如下图所示)。

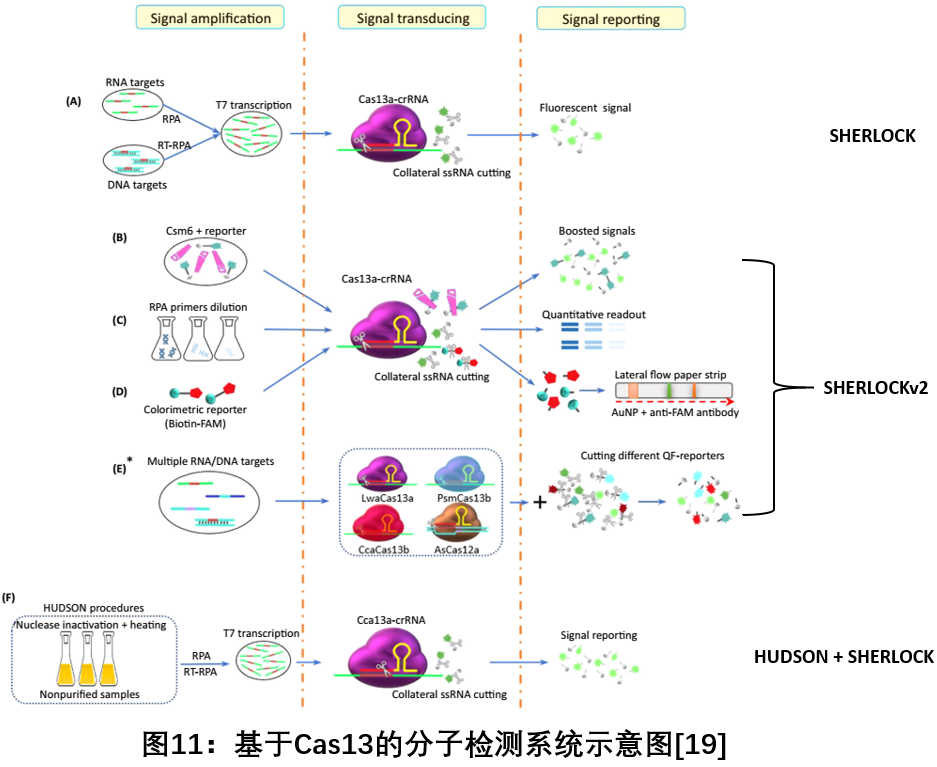
1. 基于Cas13的分子检测系统
自2017年张锋团队利用Cas13a (以前称为C2c2 ) 在靶识别时表现出混杂核糖核酸酶活性的“附带效应”与等温扩增相结合,建立了基于CRISPR的诊断方法( CRISPR-Dx ),一种具有同向敏感性和单碱基错配特异性的快速DNA或RNA检测技术---SHERLOCK(Specific High-sensitivity Enzymatic Reporter unLOCKing)诊断平台,可用于检测寨卡病毒和登革病毒、细菌分离物、抗生素抗性基因、人类DNA基因型和癌症突变等[17]。之后不久,该团队对SHERLOCK诊断平台进行一系列优化,成功开发了SHERLOCKv2系统,该系统集成了四种正交的CRISPR-Cas酶,是目前唯一可以实现同时检测多个靶向位点的检测系统,突出其多向、便携、快速和定量核酸检测平台的潜力[18]。同时,他们也进一步开发了HUDSON(heating unextracted diagnostic samples to obliterate nucleases)系统,与SHERLOCK相辅相成,用于直接从体液中检测病毒,能够在不用任何仪器的情况下在不到2小时内直接从患者样品中进行检测,极大地提高了分子检测的便利性[19]。
2. 基于Cas12的分子检测系统
l Jennifer A. Doudna团队也通过将Cas12a (CpfI)与分子信号枪相结合实现了灵敏而又准确的DNA检测。这种方法被称作DETECTR(DNA Endonuclease Targeted CRISPR Trans Reporter)。这一方法的基础在于发现Cas12a在靶向到DNA靶标后具有切割附近所有单链DNA的功能,开发人员利用一种荧光分子通过单链DNA与另一种抑制这一荧光分子发光的抑制分子连接在一起。在Cas12a切割单链DNA后将会移除一直分子,从而让这一荧光分子发光。利用这一技术,他们成功对致癌性HPV病人样品进行了正确的判断[20]。同时我国王金课题组在3月12日的Cell Research杂志上也发表了题为CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA的研究论文,同前期Jennifer Doudna课题组发表的的研究结果类似,系统地研究了Cas12a对于靶标ssDNA和非靶标ssDNA的切割特性[21]。
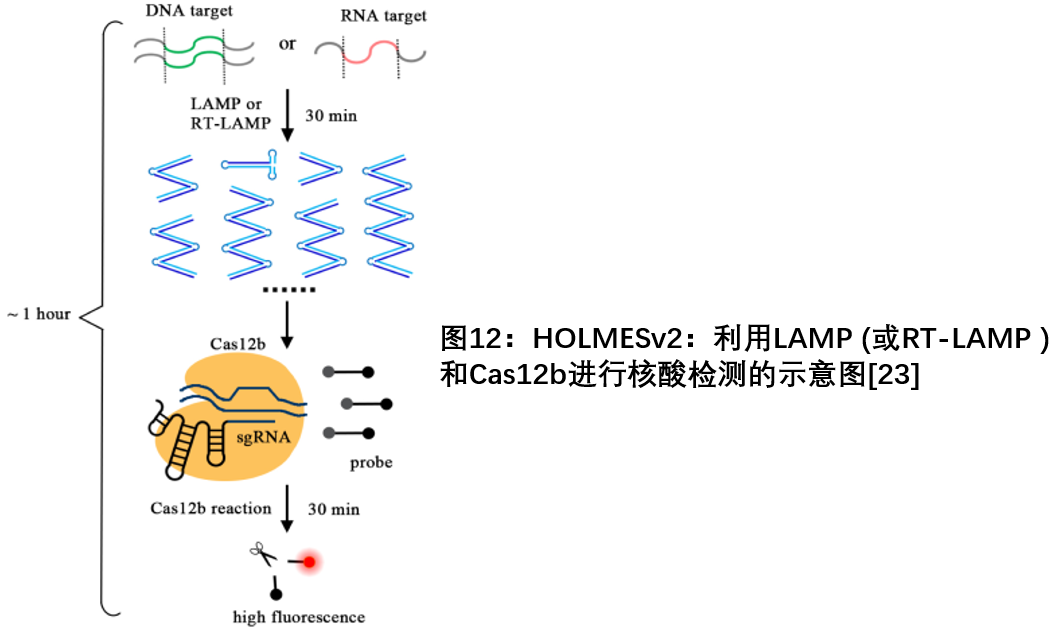
l 此外,上海吐露港的研究团队在2017年基于对侧链单链DNA具有反式切割活性的Cas12a开发的一种高效、灵敏和特异的检测DNA、RNA病毒和人的SNP的平台HOLMES (one-HOur Low-cost Multipurpose highly Efficient System) [22]。随后于2018年年底,该团队将同样具有ssDNA反式切割活性的嗜热性Cas12b与LAMP介导的等温扩增( LAMP )相结合,创建了改进版的HOLMESv2。在HOLMESv2中,LAMP扩增和Cas12b反式切割可以整合到一个恒温的一步系统中,因此为核酸检测带来了极大的便利。同时他们还简化了HOLMESv2中的RNA检测程序,使用RNA依赖性DNA聚合酶进行扩增,因此省略了额外的逆转录步骤,这是迄今为止最简单的用于RNA检测的CRISPR生物传感系统[23]。
小结:
综上为大家介绍了CRISPR/Cas9在2018年重大机制进展和诸多方面的应用,可以看到基础科学家已经在利用CRISPR-Cas9技术理解和操纵生物学方面取得了巨大进展,在临床上,我们可以期待基因疾病的新疗法(如利用Cas9基因编辑或CRISPRi/a)和新的诊断技术。我们能够利用大自然的技术工具箱,通过进化改造更多高效且安全的基因编辑工具为我们所用。新的CRISPR系统或许就隐藏在我们周围生物体的基因组中,未来可能会出现更多具有新的机制和功能的CRISPR衍生物,将会为人类提供强大的突破性技术。
参考文献:
[1] Bester, Assaf C., et al. "An Integrated Genome-wide CRISPRa Approach to Functionalize lncRNAs in Drug Resistance." Cell173.3(2018):649-664.e20.
[2] Sadhu, M. J., et al. "Highly parallel genome variant engineering with CRISPR-Cas9. " Nature Genetics50.4(2017):510.
[3] Guo, Xiaoge, et al. "High-throughput creation and functional profiling of DNA sequence variant libraries using CRISPR–Cas9 in yeast." Nature Biotechnology (2018).
[4] Bao, Zehua, et al. "Genome-scale engineering of Saccharomyces cerevisiae with single-nucleotide precision." Nature Biotechnology (2018).
[5] Roy, K. R., et al. "Multiplexed precision genome editing with trackable genomic barcodes in yeast." Nature Biotechnology (2018).
[6] Horlbeck, Max A., et al. "Mapping the Genetic Landscape of Human Cells." Cell (2018): S0092867418307359-.
[7] Halperin, Shakked O., et al. "CRISPR-guided DNA polymerases enable diversification of all nucleotides in a tunable window." Nature (2018).
[8] Wroblewska, A, et al. "Protein Barcodes Enable High-Dimensional Single-Cell CRISPR Screens." Cell175.4(2018):1141.
[9] Liu, Ying, et al. "Genome-wide screening for functional long noncoding RNAs in human cells by Cas9 targeting of splice sites." Nature Biotechnology (2018).
[10] Shifrut E, et al. Genome-wide CRISPR Screens in Primary Human T Cells Reveal Key Regulators of Immune Function. Cell. 2018 Dec 13;175(7): 1958-1971.e15.
[11] Kalhor, Reza, P. Mali, and G. M. Church. "Rapidly evolving homing CRISPR barcodes." Nature Methods 14.2(2016):195-200.
[12] Alemany, A. et al. Whole-organism clone tracing using single-cell sequencing. Nature 556, 108–112 (2018)
[13] Spanjaard, Bastiaan, et al. "Simultaneous lineage tracing and cell-type identification using CRISPR–Cas9-induced genetic scars." Nature Biotechnology (2018).
[14] Raj, Bushra, et al. "Simultaneous single-cell profiling of lineages and cell types in the vertebrate brain." Nature Biotechnology (2018).
[15] Reza, Kalhor, et al. "Developmental barcoding of whole mouse via homing CRISPR." Science (2018): eaat9804-.
[16] YiLi et al., CRISPR/Cas Systems towards Next-Generation Biosensing. Trends Biotechnol (2018).
[17] Gootenberg, Jonathan S., et al. "Nucleic acid detection with CRISPR-Cas13a/C2c2." Science 356. 6336 (2017): 438-442.
[18] Myhrvold, Cameron, et al. "Field-deployable viral diagnostics using CRISPR-Cas13." Science 360.6387(2018):444-448.
[19] Gootenberg, Jonathan S., et al. "Multiplexed and portable nucleic acid detection platform with Cas13, Cas12a, and Csm6." Science 360.6387(2018):eaaq0179.
[20] Chen, Janice S. , et al. "CRISPR-Cas12a target binding unleashes indiscriminate single-stranded DNase activity." Science (2018):eaar6245.
[21] Li, Shi Yuan, et al. "CRISPR-Cas12a has both cis- and trans-cleavage activities on single-stranded DNA." Cell Research (2018).
[22] Cheng, Q.X. et al. Shanghai Tolo Biotech Co.,Ltd. An application of a Cas protein, and a method and kit for detecting a target nucleic acid molecule, (2017) CN107488710A
[23] L. Li, S. Li, J. Wang, CRISPR-Cas12b-assisted nucleic acid detection platform. bioRxiv, (2018).